주성분 분석 (PCA)
주성분 분석 (Principal component analysis (PCA))은 특성들이 통계적으로 상관관계가 없도록 데이터셋을 회전시키는 기술입니다.
PCA는 원래의 변수들 사이의 겹치는 정보를 제거함으로써 변수를 줄입니다.
원래의 변수들이 선형 결합된 새로운 변수를 만듭니다.
선형 결합은 상관관계가 없습니다. 또한 소수의 원본 조합에는 대부분의 원래 정보가 포함되어 있습니다.
새로운 변수를 주성분이라고 부릅니다.
PCA : 데이터의 공분산 행렬의 고유분해
고유 값이 가장 큰 고유벡터를 찾습니다.
고유 벡터 - 주성분
고유 값 - 주성분에 의해 설명된 분산
입력 : $X_{1}, X_{2}, ..., X_{p}$ (원래의 p 변수)
데이터에서 평균을 빼서 중심을 원점에 맞춰줍니다.
스케일 효과를 제거하기 위해 각 변수를 정규화합니다.
출력 : $Z_{1}, Z_{2}, ..., Z_{p}$ (원래 변수의 선형 결합)
$Z_{1} = u_{11}X_{1} + u_{12}X_{2} + ... + u_{1p}X_{p}, ...$
모든 Z 변수의 쌍은 상관관계가 0입니다.
분산 순대로 Z들을 나열합니다. ($Z_{1}$은 제일 큰, $Z_{p}$는 제일 작은)
보통 처음 몇 개의 Z 변수에 대부분의 정보가 포함됩니다.
sample set mean : $\bar{\mathbf{x}} = \frac{1}{N} \sum_{n=1}^{N} \mathbf{x_{n}}$
variance of the projected data : $\frac{1}{N} \sum_{n=1}^{N} \left \{ \mathbf{u_{1}^{T}}\mathbf{x_{n} - \mathbf{u_{1}}^{T} \mathbf{\bar{x}}} \right \}^{2} = \mathbf{u_{1}}^{T} \mathbf{S} \mathbf{u_{1}}$
data covariacne matrix : $\mathbf{S} = \frac{1}{N} \sum_{n=1}^{N} (x_{n} - \bar{x})(x_{n} - \bar{x})^{T}$
$\mathbf{u_{1}}^{T} \mathbf{S} \mathbf{u_{1}} + \lambda_{1} (1 - \mathbf{u_{1}^{T}\mathbf{u_{1}}})$
$\mathbf{S} \mathbf{u_{1}} = \lambda_{1} \mathbf{u_{1}}$
$\mathbf{u_{1}}^{T} \mathbf{S} \mathbf{u_{1}} = \lambda_{1}$
의사 코드 (Psudocode)
목표 : 분산을 유지하는 r-차원 투영을 찾습니다.
1. 훈련 세트 $D = \left \{ \mathbf{x_{1}, ..., \mathbf{x_{N}}} \right \}$에서 원본 데이터 포인트의 공분산 행렬 $\mathbf{S}$을 계산합니다.
2. $\mathbf{S}$의 고유 값과 고유벡터를 계산합니다.
3. top r 고유벡터 $\mathbf{u_{1}}, ..., \mathbf{u_{r}}$를 선택합니다.
4. 이들에 의해 합쳐진 부분공간에 각 데이터 포인트 $\mathbf{x_{n}}$를 투영합니다.
$\mathbf{Z_{n}} = (z_{1}, ..., z_{r})$, $z_{ni} = \mathbf{u}^{T}_{i} \mathbf{x_{n}}$

예시 : 2차원 데이터셋을 사용하여 PCA 효과를 나타냅니다.
원래의 데이터를 주성분 1과 2를 각각 x축과 y축에 나란하도록 회전했습니다.
주성분 1은 분산이 가장 큽니다.
첫 번쨰 주성분과 직교하는 주성분 2를 찾습니다. 남아 있는 분산 중 가장 큰 것을 고려합니다.

유방암 데이터셋 - PCA
from sklearn.datasets import load_breast_cancer
cancer = load_breast_cancer()
유방암 데이터셋을 사용합니다.
from sklearn.model_selection import train_test_split
X_train, X_test, y_train, y_test = train_test_split(
cancer.data, cancer.target, random_state=42)
훈련 세트와 테스트 세트로 나눠줍니다.
from sklearn.preprocessing import StandardScaler
scaler = StandardScaler()
scaler.fit(X_train)
X_train_scaled = scaler.transform(X_train)
X_test_scaled = scaler.transform(X_test)
데이터 스케일링을 해줍니다.
from sklearn.decomposition import PCA
pca = PCA(n_components=2)
PCA 객체를 생성하고 주성분 개수를 2로 설정합니다.
pca.fit(X_train_scaled)
모델을 만듭니다.
X_train_pca=pca.transform(X_train_scaled)
X_test_pca=pca.transform(X_test_scaled)
transform 메서드를 호출해 데이터를 회전시키고 차원을 축소합니다.
print("Original shape: {}".format(str(X_train_scaled.shape)))
print("Reduced shape: {}".format(str(X_train_pca.shape)))
데이터 형태가 2 개로 축소된 것을 볼 수 있습니다.
X_test_scaled_pd = pd.DataFrame(X_test_scaled)
X_test_scaled_pd.head()
원본 데이터 (X_test)scaled) 입니다.
pca.components_pd = pd.DataFrame(pca.components_)
pca.components_pd
components_의 각 행은 주성분 하나씩을 나타내며 중요도에 따라 정렬되어 있습니다.
열은 원본 데이터의 특성에 대응하는 겂입니다.
X_test_pca_pd = pd.DataFrame(X_test_pca)
X_test_pca_pd.head()
변환된 데이터 (X_test_pca) 입니다.

처음 두 개의 주성분을 사용해 그린 유방암 데이터셋의 산점도입니다.
PC의 갯수 선택하기
데이터 시각화를 위해 차원을 줄이는 경우 일밙거으로 2 또는 3 정도로 차원을 줄이면 됩니다.
그렇지 않은 경우에는 충분히 큰 분산의 비율 (예를 들어 95%)에 준하는 차원의 수를 선택하는 것을 선호합니다.
예시 : 차원 수의 함수로 설명된 분산

pca = PCA(n_components=0.95)
pca.fit(X_train_scaled)
n_components를 float으로 지정하는 경우, 원하는 분산 비율을 나타냅니다.
n_components=0.95로 설정했습니다.
X_train_pca = pca.transform(X_train_scaled)
print("Original shape: {}".format(str(X_train_scaled.shape)))
print("Reduced shape: {}".format(str(X_train_pca.shape)))
데이터의 형태가 10개로 축소되었습니다.
pca.components_[0]
주성분 1의 모든 특성은 위와 같습니다.
pca.explained_variance_ratio_[:10]
설명 가능한 분산 비율을 모두 더하면 0.9518이 나옵니다.
pca = PCA()
pca.fit(X_train_scaled)
차원 축소를 하지 않고 PCA를 계산할 수 있습니다. 이 경우엔 사용자가 원하는 훈련 세트의 분산의 특정 비율을 택한 후 이에 적합한 차원의 수를 계산하면 됩니다.
X_train_pca = pca.transform(X_train_scaled)
print("Original shape: {}".format(str(X_train_scaled.shape)))
print("Reduced shape: {}".format(str(X_train_pca.shape)))
데이터 형태가 축소되지 않았습니다.
pca.explained_variance_ratio_[:30]
설명된 분산의 비는 위와 같습니다.
sum(pca.explained_variance_ratio_[:10])
10번 째에서 끊으면 아까와 동일하게 0.9518이 나오는 것을 알 수 있습니다.
주성분 1, 2를 히트맵을 사용하여 시각화하겠습니다.
모든 특성 사이에 공통의 상호관계가 있습니다.
화살표 방향은 의미가 없습니다.
모든 특성이 섞여 있기 때문에 축이 가지는 의미를 설명하기는 쉽지 않습니다.
pca.components_[:2]
plt.matshow(pca.components_, cmap='viridis')
plt.yticks([0, 1], ["First component", "Second component"])
plt.colorbar()
plt.xticks(range(len(cancer.feature_names)),
cancer.feature_names, rotation=60, ha='left')
plt.xlabel("Feature")
plt.ylabel("Principal components")
모든 성분이 섞여 있는 것을 볼 수 있습니다.
지도 학습에서의 PCA
PCA는 지도 학습을 위한 전처리 과정에서 사용됩니다.
지도 학습 알고리즘의 정확도를 개선시켜 줄 때가 있습니다.
메모리와 시간을 감소시킬 수 있습니다.
주요 단계
PCA를 훈련 데이터에 적용합니다.
얼마나 많은 PC를 사용할 것인지 결정합니다.
평가/테스트 데이터에 PC를 사용합니다.
그 결과 평가/테스트 데이터 안에 줄어든 예측 변수의 셋이 새롭게 생성됩니다.
유방암 데이터셋 - PCA in Supervised Learning
30개의 변수를 가진 본 데이터를 사용합니다.
from sklearn.datasets import load_breast_cancer
cancer = load_breast_cancer()
유방암 데이터셋을 불러옵니다.
from sklearn.model_selection import train_test_split
X_train, X_test, y_train, y_test = train_test_split(
cancer.data, cancer.target, random_state=42)
훈련과 테스트 셋으로 나눠줍니다.
from sklearn.preprocessing import StandardScaler
scaler = StandardScaler()
scaler.fit(X_train)
X_train_scaled = scaler.transform(X_train)
X_test_scaled = scaler.transform(X_test)
데이터 스케일링을 해줍니다.
from sklearn.neighbors import KNeighborsClassifier
from sklearn.metrics import accuracy_score
clf = KNeighborsClassifier(n_neighbors=3)
clf.fit(X_train_scaled, y_train)
y_train_hat = clf.predict(X_train_scaled)
print('train accuracy :', accuracy_score(y_train, y_train_hat))
y_test_hat = clf.predict(X_test_scaled)
print('test accuracy :', accuracy_score(y_test, y_test_hat))
k-최근접 이웃 분류를 사용하여 성능을 평가했습니다.
주성분이 2개인 새로운 데이터를 사용합니다.
from sklearn.decomposition import PCA
pca = PCA(n_components=2)
pca.fit(X_train_scaled)
X_train_pca=pca.transform(X_train_scaled)
X_test_pca=pca.transform(X_test_scaled)
PCA 모델을 만듭니다.
from sklearn.neighbors import KNeighborsClassifier
from sklearn.metrics import accuracy_score
clf = KNeighborsClassifier(n_neighbors=3)
clf.fit(X_train_pca, y_train)
y_train_hat = clf.predict(X_train_pca)
print('train accuracy :', accuracy_score(y_train, y_train_hat))
y_test_hat = clf.predict(X_test_pca)
print('test accuracy :', accuracy_score(y_test, y_test_hat))
본 데이터보다 훈련 정확도는 감소하였지만 테스트 정확도는 증가하여 일반화가 더 잘 되었습니다.
데이터 재구성을 위한 PCA
PCA로 차원이 감소하면 훈련 세트는 더 적은 공간을 갖게 됩니다.
PCA 투영의 역변환을 적용하여 축소 된 데이터 세트를 원래 차원으로 다시 되돌릴 수 있습니다.
재설정 $\mathbf{x_{n}} = (\mathbf{x_{n1}}, ..., \mathbf{x_{np}})$ from $\mathbf{z_{n}} = (z_{n1}, ..., z_{nr})$
$\widetilde{\mathbf{x_{n}}} = \sum_{i=1}^{r}z_{ni}\mathbf{u_{i}}$
이것은 본 데이터로 완벽하게 돌아가진 않더라도 꽤 가까운 수치를 자랑합니다.

pca = PCA()
pca.fit(X_train_scaled)
n_components를 정해주지 않고 모델을 만듭니다.
X_test_pca = pca.transform(X_test_scaled)
X_test_rec = pca.inverse_transform(X_test_pca)
본 데이터와 재구성된 데이터를 만들었습니다.
X_test_scaled_pd = pd.DataFrame(X_test_scaled)
X_test_scaled_pd.head()
본 데이터 (X_test_scaled) 입니다.
X_test_rec_pd = pd.DataFrame(X_test_rec)
X_test_rec_pd.head()
재구성된 데이터 (X_test_rec) 입니다.
완전히 똑같습니다.
pca = PCA(n_components=15)
pca.fit(X_train_scaled)
n_components=15로 주성분 개수를 제한했습니다. 50% compression
X_test_pca = pca.transform(X_test_scaled)
X_test_rec = pca.inverse_transform(X_test_pca)
본 데이터와 재구성된 데이터를 만들었습니다.
X_test_scaled_pd = pd.DataFrame(X_test_scaled)
X_test_scaled_pd.head()
본 데이터 (X_test_scaled) 입니다.
X_test_rec_pd = pd.DataFrame(X_test_rec)
X_test_rec_pd.head()
주성분 개수를 제한 했더니 수치가 조금 달라졌습니다.
pca = PCA(n_components=3)
pca.fit(X_train_scaled)
n_componenst=3으로 설정했습니다. 90% compression
X_test_pca = pca.transform(X_test_scaled)
X_test_rec = pca.inverse_transform(X_test_pca)
본 데이터와 재구성된 데이터를 만들었습니다.
X_test_scaled_pd = pd.DataFrame(X_test_scaled)
X_test_scaled_pd.head()
본 데이터 (X_test_scaled) 입니다.
X_test_rec_pd = pd.DataFrame(X_test_rec)
X_test_rec_pd.head()
역시 수치가 달라졌습니다.
주성분 개수에 따라 재구성된 데이터의 수치가 차이가 있습니다.
얼굴 데이터셋 - PCA for Data Compression

얼굴 데이터셋의 주성분 중 처음 15개입니다.
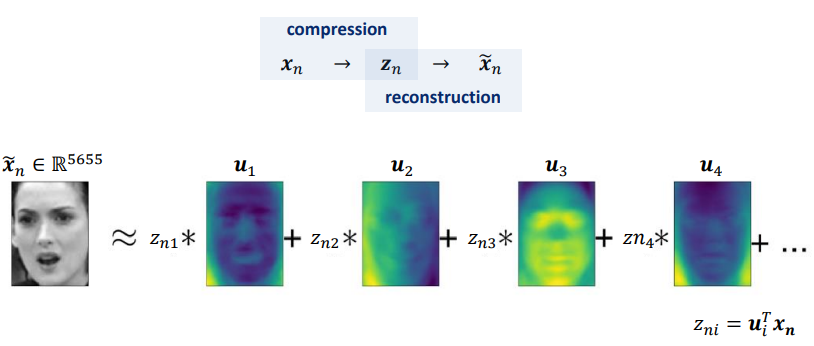
Schematic view of PCA는 테스트 포인트를 주성분의 가중치 합으로 나타내는 데 필요한 수치를 찾는 것입니다.

주성분의 숫자를 늘려가며 얼굴 이미지를 재구성했습니다.
파이썬 라이브러리를 활용한 머신러닝 책과 성균관대학교 강석호 교수님 수업 내용을 바탕으로 요약 작성되었습니다.
'파이썬 라이브러리를 활용한 머신러닝' 카테고리의 다른 글
| K-Means Clustering (0) | 2019.11.25 |
|---|---|
| t-distributed Stochastic Neighbor Embedding (0) | 2019.11.19 |
| Unsupervised Learning & Data Preprocessing (0) | 2019.11.15 |
| Uncertainty Estimates from Classifiers & Summary and Outlook (0) | 2019.11.15 |
| Neural Networks (0) | 2019.11.14 |




댓글